FASTA (.fasta,.fa,.fna,.fsa,.mpfa)
予備知識

-
- MIMEタイプ:chemical/seq-aa-fasta,chemical/seq-na-fasta
- FASTA分子生物学形式.
- DNAと蛋白質の配列の保管と交換の標準形式.
- プレーンテキスト形式.
- 核酸と蛋白質の配列を文字列で保管する.
- メタ情報を表すために多くの表記方法が使われている.
- 1988年にWilliam Pearson とDavid LipmanによりFASTAシーケンスアライメントソフトウエアの一部として開発された.
ImportとExport
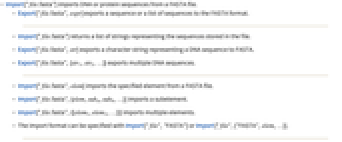
- Import["file.fasta"]はDNAまたは蛋白質配列をFASTAファイルよりインポートする.
- Export["file.fasta",expr] は配列または配列のリストをFASTA形式にエキスポートする.
- Import["file.fasta"]はファイルに保管されている配列を表す文字列のリストを返す.
- Export["file.fasta",str]はDNAの配列を表している文字列をFASTAにエキスポートする.
- Export["file.fasta",{str1,str2,…}]は複数のDNA配列をエキスポートする.
- Import["file.fasta",elem]は指定された要素をFASTAファイルよりインポートする.
- Import["file.fasta",{elem,suba,subb,…}]はサブ要素をインポートする.
- Import["file.fasta",{{elem1,elem2,…}}]は複数の要素をインポートする.
- インポート形式はImport["file","FASTA"]あるいはImport["file",{"FASTA",elem,…}]として指定できる.
- Export["file.fasta",expr,elem]はexpr が要素,elem を指定するものとして扱うことにより,FASTAファイルを作成する.
- Export["file.fasta",{expr1,expr2,…},{{elem1,elem2,…}}]では,それぞれのexpri が対応するelemiを指定するものとして扱われる.
- Export["file.fasta",expr,opt1->val1,…]はexpr を特別の値を持つオプションが指定されたものとしてエキスポートする.
- Export["file.fasta",{elem1->expr1,elem2->expr2,…},"Rules"]は規則を使い,エキスポートされる要素を指定する.
- 一般的な情報は,以下の関数ページを参照のこと.
-
Import, Export ファイルからインポートする,あるいはファイルへエキスポートする CloudImport, CloudExport クラウドオブジェクトからインポートする,あるいはクラウドオブジェクトへエキスポートする ImportString, ExportString 文字列からインポートする,あるいは文字列へエキスポートする ImportByteArray, ExportByteArray バイト配列からインポートする,あるいはバイト配列へエキスポートする
Import要素



- 一般的なImport要素:
-
"Elements" ファイル中の有効な要素とオプションのリスト "Summary" ファイルの概要 "Rules" 使用可能なすべての要素の規則のリスト - データ表現要素:
-
"Header" ヘッダラインそのもの "Sequence" 文字列のリストとしてのDNAまたは蛋白質の配列 "Plaintext" 書式化されたテキストとしての配列 - Importはデフォルトでは"Sequence"要素をFASTA形式で使う.
- 追加的データ要素:
-
"Data" リストでの"Header"と"Sequence"要素の組合せ "LabeledData" この保管されている個々の配列のための規則のリスト - ヘッダラインメタ情報:
-
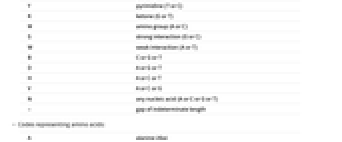
"Accession" それぞれの配列のNCBI アクセッション番号 "Description" それぞれの配列で位置説明のテキスト "GenBankID" GenBankデータベース識別子 "Length" 個々の配列の長さを表す整数のリスト - Wolfram言語はIUB/IUPAC規定を核酸の省略形に使う:
-
A アデノシン C シチジン G グアニン T チミジン U ウラシル R プリン (GまたはA) Y ピリミジン (TまたはC) K ケトン (GまたはT) M アミノ基 (AまたはC) S 強い結合 (GまたはC) W 弱い結合 (AまたはT) B CまたはGまたはT D AまたはGまたはT H AまたはCまたはT V AまたはCまたはG N 任意の核酸 (AまたはCまたはGまたはT) - 不明な長さのギャップ - アミノ酸を表すコード:
-
A アラニン(Ala) B アスパラギン酸 もしくは アスパラギン C システイン(Cys) D アスパラギン酸(Asp) E グルタミン酸(Glu) F フェニルアラニン(Phe) G グリシン(Gly) H ヒスチジン(His) I イソロイシン(Ile) K リシン(Lys) L ロイシン(Leu) M メチオニン(Met) N アスパラギン(Asn) P プロリン(Pro) Q グルタミン(Gln) R アルギニン(Arg) S セリン(Ser) T スレオニン(Thr) U セレノシステイン V バリン(Val) W トリプトファン(Trp) Y チロシン(Tyr) Z グルタミン酸 もしくは グルタミン X 任意アミノ酸 * 翻訳終止 - 不明な長さのギャップ
オプション

- Importオプション:
-
"HeaderFormat" Automatic ヘッダのフォーマットを指定 "ToUpperCase" True 配列を大文字にするかどうか - ImportはFASTA形式で使われるヘッダフォーマットの一般的な指定のものの大規模な組込みライブラリを使う.
- "HeaderFormat"を文字列またはメタ情報要素のリストに設定することにより,任意のヘッダラインフォーマットをImportで指定することができる.
- "HeaderFormat"->{"gi","DatabaseIndex","gb","Accession","","Description"}はNCBI FASTAファイルの典型的な設定である.
- 高度なExportオプション:
-
"LineWidth" 70 行の最大文字数 "ToUpperCase" True 配列を大文字にするかどうか