MMJSON (.json)
予備知識
-
- 3D分子モデルファイル
- 化学情報アプリケーションやWeb上で,分子モデルの保存と交換をするために使用される.
- Protein Data Bank JapanがPDB形式の代替として使用する
- Derived from the JSONファイル形式から派生したものである.
- プレーンテキスト形式.
- タンパク質や核酸などの大きな生物学的分子の構造情報を格納する.
ImportとExport
- Import["file.json","MMJSON"] はmmJSONファイルを読み込み,生体分子の記号表現を返す.
- Wolfram言語は高分子のためのさまざまな3次元レンダリングのスタイルを提供する.
- Export["file.json",{biomol,"MMJSON"}] は生体分子からMMJSONファイルを作成する.
- Import["file.json","MMJSON"] はBioMoleculeオブジェクトを返す.
- Import["file.json",{"MMJSON",elem}] は指定された要素をmmJSONファイルからインポートする.
- Import["file.json",{"MMJSON",elem,suba,subb,…}]はサブ要素をインポートする.
- Import["file.json",{"MMJSON"{elem1,elem2,…}}]は複数の要素をインポートする.
- Export["file.json",biomol]はBioMolecule biomolをエキスポートする.
- 一般的な情報は,以下の関数ページを参照のこと.
-
Import, Export ファイルからインポートする,あるいはファイルへエキスポートする CloudImport, CloudExport クラウドオブジェクトからインポートする,あるいはクラウドオブジェクトへエキスポートする ImportString, ExportString 文字列からインポートする,あるいは文字列へエキスポートする ImportByteArray, ExportByteArray バイト配列からインポートする,あるいはバイト配列へエキスポートする
Import要素



- 一般的なImport要素:
-
"Elements" ファイル中の有効な要素とオプションのリスト "Summary" ファイルの概要 "Rules" 使用可能なすべての要素の規則のリスト - データ要素:
-
"BioMolecule" 高分子モデルの記号表現 "Molecule" 分子モデルの記号表現 - Import と Export はデフォルトでmmJSON形式に"BioMolecule" 要素を使用する.
- BioMolecule オブジェクトには鎖と残基についての情報および原子の種類と座標の情報が含まれる.Moleculeオブジェクトは原子間の結合を割当て,残基や鎖のラベルなどのメタ情報を破棄する.
- グラフィックス要素:
-
"Graphics3D" Graphics3Dオブジェクトとして描画されたmmJSONファイル - データ表現要素:
-
"Residues" 3文字短縮形の配列としての残基列 "Sequence" 文字列のリストとして与えられる残基列 "ResidueAtoms" 残留原子のリスト "ResidueChainLabels" 鎖ラベルのリスト "ResidueRoles" 残留原子の機能的役割 "ResidueCoordinates" 残留原子の3次元座標(オングストローム単位) "Resolution" モデル座標の空間解像度(オングストローム単位) "AdditionalAtoms" 鎖の構成要素ではない原子 "AdditionalCoordinates" 付加原子の3D座標 "AdditionalResidues" 3文字省略形の配列としての追加的な残基列 "SecondaryStructure" 鎖の大規模な構造を表現する規則 "VertexCoordinates" オングストロームで与えられる原子座標 "VertexTypes" 分子を構成しているすべての原子あるいはグループ(通常化学要素のリストとして与えられる) - Wolfram言語はアミノ酸残基に標準のIUB/IUPAC短縮形を使用する.
- 同じ分子の複数の3次元モデルを表現するmmJSONファイルをインポートする際には,全モデルの形状を読み込むために以下のImport 要素を使うことができる:
-
"ResidueCoordinatesList" それぞれのモデルについての残基座標 "AdditionalCoordinatesList" それぞれのモデルについての付加原子の3次元座標 "VertexCoordinatesList" それぞれのモデルについての原子座標(オングストローム単位) - メタ情報要素:
-
"Authors" ファイルで参照される著者情報 "DepositionDate" いつファイルがデータベースに加えられたか "PDBClassification" ファイルヘッダからのPDB分類 "PDBID PDBの構造識別文字列 "References" 規則のリストとして与えられる書誌参照 "Title" ドキュメントタイトル
オプション

- "BioMolecule" インポート要素は以下のオプションを取る:
-
"DetectSecondaryStructure" Automatic - 残基のリストをスキャンしてヘリックスとシートを検出するかどうか
- "Graphics3D" インポート要素はBioMoleculePlot3Dと同じオプションを取る.
- レンダリングスタイルを選択する:
-
PlotTheme "Ribbons" 可視化の手法を指定する - サポートされるプロットテーマは以下を含む:
-
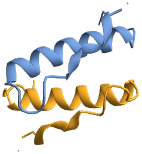
"Ribbons" ポリマー鎖をリボンとして表示する 
"Backbone" ポリマー鎖をリボンとして表示する 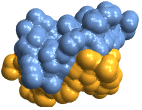
"SolventAccessibleSurface" 溶剤が浸透しやすい表面 
"GaussianSurface" ガウス表面 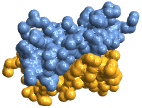
"VanDerWaalsSurface" ファンデルワールス表面 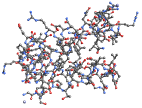
"BallAndStick" SphereとCylinderプリミティブを使って原子と結合を表示する 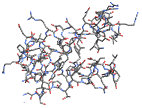
"Tubes" 原子なしのチューブとして結合を表示する 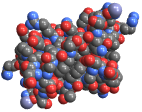
"Spacefilling" 原子はファンデルワールス半径に一致する半径の球で表される
例題
例 (3)
BioMoleculeをmmJSONファイルにエキスポートする:
使用可能な全てのImport要素の名前を表示する:
MoleculeオブジェクトをmmJSONにエキスポートする:
ファイルを Graphics3Dとしてインポートする: