PDB (.pdb)
予備知識

-
- MIMEタイプ:chemical/x-pdb
- Protein Data BankのPDBファイル.
- 3次元分子模型のファイル.
- 生物情報学のアプリケーション内やWeb上で,分子模型を保持したり,交換したりするのに使用される.
- PDBはProtein Data Bank(タンパク質データバンク)の頭字語である.
- プレーンテキスト形式.
- タンパク質や核酸等の大きな生体分子の構造情報を保持する.
- 化学結合の情報は保持しない.
- 1971年にBrookhaven National Laboratoryで開発された.
- Research Collaboratory for Structural Bioinformatics (RCSB)で管理されている.
ImportとExport

- Import["file.pdb"]はPDBファイルを読み取り,タンパク質の定型化したレンダリングを返す.
- Wolfram言語は高分子のためのさまざまな3次元レンダリングスタイルを提供する.
- Export["file.pdb",expr]は分子の3D模型からPDFファイルを作成する.
- Import["file.pdb"]はGraphics3Dオブジェクトを返す.
- Import["file.pdb",elem] は指定された要素をPDBファイルからインポートする.
- Import["file.pdb",{elem,suba,subb,…}]はサブ要素をインポートする.
- Import["file.pdb",{{elem1,elem2,…}}]は複数の要素をインポートする.
- インポートの形式は Import["file","PDB"] あるいは Import["file",{"PDB",elem,…}]で指定することができる.
- Export["file.pdb",{elem1->expr1,elem2->expr2,…}]はエキスポートする要素を規則を使って指定する.
- ImportとExportについての完全な一般情報は関数ページを参照のこと.
- ImportStringとExportStringはPDB形式をサポートする.
- 一般的な情報は,以下の関数ページを参照のこと.
-
Import, Export ファイルからインポートする,あるいはファイルへエキスポートする CloudImport, CloudExport クラウドオブジェクトからインポートする,あるいはクラウドオブジェクトへエキスポートする ImportString, ExportString 文字列からインポートする,あるいは文字列へエキスポートする ImportByteArray, ExportByteArray バイト配列からインポートする,あるいはバイト配列へエキスポートする
Import要素
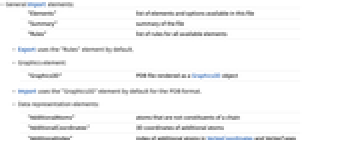



- 一般的なImport要素:
-
"Elements" ファイル中の有効な要素とオプションのリスト "Summary" ファイルの概要 "Rules" 使用可能なすべての要素の規則のリスト - Exportはデフォルトでは"Rules"要素を使用する.
- グラフィックス要素:
-
"Graphics3D" Graphics3Dオブジェクトとして描画されるPDBファイル - Importは"Graphics3D"要素をデフォルトでPDB形式に使う.
- データ表現要素:
-
"AdditionalAtoms" 鎖の構成要素ではない原子 "AdditionalCoordinates" 付加原子の3D座標 "AdditionalIndex" VertexCoordinatesとVertexTypesの追加の原子の指標 "AdditionalResidues" 3文字短縮形の配列として与えられる追加的な残基列 "Molecule" 分子モデルの記号表現 "ResidueAtoms" 残留原子のリスト "ResidueChainLabels" 鎖ラベルのリスト "ResidueCoordinates" 残留原子の3D座標 "ResidueIndex" VertexCoordinatesとVertexTypesの残留原子の指標 "ResidueRoles" 残留原子の機能的役割 "Residues" 3文字短縮形の配列として与えられる残基列 "Resolution" 模型座標の空間分解能(ピコメートル) "SecondaryStructure" 鎖の大規模な構造を表現する規則 "Sequence" 文字列のリストとして与えられる残基列 "VertexCoordinates" 原子座標(通常ピコメートルで与えられる) "VertexTypes" 分子を構成しているすべての原子あるいはグループ(通常化学要素の短縮形のリストで与えられる) - 1つかそれ以上の残基が欠けている不完全な鎖をPDBから読み取る場合は,Wolfram言語はそれを個々の部分鎖の列として表示する.
- Wolfram言語はアミノ酸残基に標準のIUB/IUPAC短縮形を使う:
-
A アラニン (Ala) C システイン (Cys) D アスパラギン酸 (Asp) E グルタミン酸 (Glu) F フェニルアラニン (Phe) G グリシン (Gly) H ヒスチジン (His) I イソロイシン (Ile) K リジン (Lys) L ロイシン (Leu) M メチオニン (Met) N アスパラギン (Asn) P プロリン (Pro) Q グルタミン (Gln) R アルギニン (Arg) S セリン (Ser) T トレオニン (Thr) V バリン (Val) W トリプトファン (Trp) Y チロシン (Tyr) X 詳細不明あるいは未知のアミノ酸 (Unk) - 以下の短縮形は核酸を表示するのに使われる:
-
A アデノシン C シチジン G グアノシン I イノシン T チミジン U ウリジン X 詳細不明あるいは未知の核酸 - 同じ分子の複数3次元模型を表現するPDBファイルをインポートする場合には,すべての模型の形状を読み取るのに次のImport要素を使うことができる:
-
"ResidueCoordinatesList" それぞれの模型についての残基座標 "AdditionalCoordinatesList" それぞれの模型についての付加原子の3次元座標 "VertexCoordinatesList" それぞれの模型についての原子座標(通常ピコメートルで与えられる) - メタ情報要素:
-
"Author" ファイルで参照される著者情報 "Comments" ファイル内に保持されたユーザのコメント(文字列のリストとしてあたえられる) "DepositionDate" いつファイルがデータベースに加えられたか "Organism" タンパク質が起る有機体 "PDBClassification" ファイルヘッダからのPDB分類 "PDBID PDBの構造識別文字列 "References" 書誌参照(規則のリストとして与えられる) "Title" ドキュメントタイトル
オプション

- 一般的なレンダリングオプション:
-
ImageSize Automatic 表示するグラフィックスの全体的な大きさを指定する Background White どの背景色を使うかを指定する ColorFunction Automatic 二次構造の可視化の色付けを決定するために適用する関数 ViewPoint Automatic 3次元模型を見る空間内の視点 - デフォルト設定のViewPoint->Automaticで,Wolfram言語はインポートされた分子模型を見るのに最適な角度を自動的に計算する.
- レンダリングスタイルを選択する:
-
"Rendering" "Structure" 可視化メソッドを指定する - "Rendering"に可能な設定:
-
"BallAndStick" 原子と結合を玉と棒の模型として表示する "Structure" タンパク質骨格の定型化されたレンダリング "Spacefilling" 重なり合う球として表示された原子 "Wireframe" 線として描画された結合
例題
例 (6)
大きなPDBファイルをRCSB Protein Data BankのWebサイトからインポートする:
より小さなサンプルファイル内で使用可能なImport 要素を示す:
構造をMoleculeオブジェクトとしてインポートする:
それぞれの残基について標準色を使って,同じタンパク質を示す:
以下ではサンプルファイルを玉と棒のグラフィックスでインポートする:
以下ではサンプルファイルをインポートして,原子を空間充填の球体として描画する:
PDBファイルからすべてのデータを読み込み,それをPDBにもう一度エキスポートする:
MOLファイルから簡単な3D模型をインポートしてそれをPDBにエキスポートする:
結果のPDBファイルを3Dグラフィックスとしてインポートする:
MoleculeオブジェクトをPDB文字列としてエキスポートする: