BioSequence
BioSequence[type,"seq"]
表示对应字符串 "seq" 的给定类别 type 的生物分子序列.
BioSequence["seq"]
从序列中推断类型(DNA,蛋白质等).
BioSequence[ent]
给出与基因或蛋白质实体 ent 相关的生物分子序列.
BioSequence[type,{chem1,chem2,…}]
给出与给定化学物列表相对应类型 type 的生物分子序列.
BioSequence[type,"seq",{bond1,bond2,…}]
表示有给定键列表的生物分子序列.
BioSequence["HybridStrand",{bioseq1,bioseq2,…},{bond1,bond2,…}]
表示由多个 motif 序列和共享主要连锁组成的序列.
BioSequence[{bioseq1,bioseq2,…},{bond1,bond2,…}]
表示仅由额外键链接的多个序列.
更多信息和选项



- 如果可能,BioSequence[…] 计算给出以下格式:
-
BioSequence[type,"seq",bonds] 基序(单一类型的单链) BioSequence["HybridStrand",{bioseq1,bioseq2,…},bonds] 混合链(多种类型的单链) BioSequence[{bioseq1,bioseq2,…},bonds] 序列集合(许多带有附加键的链) - BioSequence 使用下列字母代表每个类型 type 的分子:
-
"DNA" A, C, G, T "CircularDNA" A, C, G, T "RNA" A, C, G, U "CircularRNA" A, C, G, U "Peptide" A, C, D, E, F, G, H, I, K, L, M, N, O, P, Q, R, S, T, U, V, W, Y "CircularPeptide" A, C, D, E, F, G, H, I, K, L, M, N, O, P, Q, R, S, T, U, V, W, Y - 此表的内容可通过 "BioSequenceType" 实体的 "Alphabet" 属性 Entity["BioSequenceType","DNA"]["Alphabet"].
- 以下是 DNA (RNA) 字母对应的核苷酸:
-
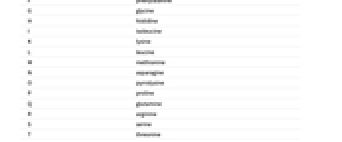
A 腺嘌呤 C 胞嘧啶 G 鸟嘌呤 T (U) 胸腺嘧啶(尿嘧啶) - 同样,以下是每个肽字母对应的氨基酸:
-
A 丙氨酸 C 半胱氨酸 D 天冬氨酸 E 谷氨酸 F 苯丙氨酸 G 甘氨酸 H 组氨酸 I 异亮氨酸 K 赖氨酸 L 白氨酸 M 甲硫氨酸 N 天冬酰胺 O 吡咯赖氨酸 P 脯氨酸 Q 谷氨酰胺 R 精氨酸 S 丝氨酸 T 苏氨酸 U 硒代半胱氨酸 V 缬氨酸 W 色氨酸 Y 酪氨酸 - 前面表格的内容可通过 "BioSequenceType" 实体的 "AlphabetRules" 属性获得,例如通过 Entity["BioSequenceType","DNA"]["AlphabetRules"].
- "Peptide" 和 "CircularPeptide" 类型还允许句号或星号(. 或 *),用于代表在生物分子翻译发生时在何处停止.
- 此外,类型 type 可以设置为 None,用于代表没有给定化学物意义的类别序列.
- BioSequence 还允许代表多个可能化学物的退化性字母.
- 允许的 DNA 和 RNA 的退化性字母包括:
-
B C,G 或 T/U (非 A) D A,G 或 T/U (非 C) H A,C 或 T/U (非 G) K G 或 T/U (酮) M A 或 C (氨基) N A,C,G 或 T/U (任何字母) R A 或 G (嘌呤) S C 或 G (强) V A,C 或 G (非 T) W A 或 T/U (弱) Y C 或 T/U (嘧啶) - 允许的关于肽的退化性字母包括:
-
B D 或 N J I 或 L X A, C, D, E, F, G, H, I, K, L, M, N, O, P, Q, R, S, T, U, V, W, Y Z E 或 Q - 前面表格的内容可通过 "BioSequenceType" 实体的 "DegenerateLetterRules" 属性获得,例如通过 Entity["BioSequenceType","DNA"]["DegenerateLetterRules"].
- 在提供了类型和长度后,下列字母看用作任意字母:
-
"DNA" 或 "CircularDNA" N "RNA" 或 "CircularRNA" N "Peptide" 或 "CircularPeptide" X - BioSequence 接受标准缩写代替序列字母.
- DNA 碱基的可能缩写包括:
-
"dAdo" A "dCyd" C "dGuo" G "dNuc" N "dPuo" R "dThd" T "dPyd" Y - RNA 碱基的可能缩写包括:
-
"Ado" A "Cyd" C "Guo" G "Nuc" N "Puo" R "Urd" U "Pyd" Y - 氨基酸的可能缩写包括:
-
"Ala" A "Asx" B "Cys" C "Asp" D "Glu" E "Phe" F "Gly" G "His" H "Ile" I "Xle" J "Lys" K "Leu" L "Met" M "Asn" N "Pyl" O "Pro" P "Gln" Q "Arg" R "Ser" S "Thr" T "Sec" U "Val" V "Trp" W "Xaa" X "Tyr" Y "Glx" Z - 除了序列暗示的连接之外,BioSequence 字母还可以通过附加的 Bond 条目连接.
- 以 Bond[{i,j},type] 形式指定的键通过 type 类型的键连接对应于字符串位置 i 和 j 的化学物质. 例如,连接 DNA 序列 "ACCT" 中 "A" 和 "T" 的氢键可以表示为 BioSequence["DNA","ACCT",{Bond[{1,4},"MultiHydrogen"]}].
- 序列级别的单键可以代表分子级别的多个键. 在前面的例子中,"A" 和 "T" 之间的 Bond 代表分子水平上的两个氢键.
- 在混合链中,形式为 Bond[{{i1,i2},{j1,j2}},type] 的键通过指定 type 的键连接分别在位置 i2 和 j2 处具有索引 i1 和 j1 的基序链. 例如,连接 DNA/RNA 杂交序列 {"ACC","CCU"} 中 "A" 和 "U" 的氢键可以表示为 BioSequence["HybridStrand",{"ACC","CCU"},{Bond[{{1,2},{2,3}},"MultiHydrogen"]}].
- 在序列集合中,形式为 Bond[{{i1,i2,i3},{j1,j2, j3}},type] 的键通过类型为 type 的键连接分别在位置 i3 和 j3 处具有索引 {i1,i2} 和 {j1,j2} 的基序链.
- 如果在序列集合层面连接基序链,则可以使用 {i1,1,i3} 或 {i1,i3}. 例如,给定两个 DNA 序列 "CAC" 和 "CTC",连接第一个序列的 "A" 和第二个序列的 "T" 的氢键可以表示为 BioSequence[{"CAC","CTC"},Bond[{{1,1,2},{2,1,2}},"MultiHydrogen"]] 或 BioSequence[{"CAC","CTC"},Bond[{{1,2},{2,2}},"MultiHydrogen"]] .
- 对于序列集合中的混合链,需要所有索引. 例如,假设 DNA/RNA 混合序列 {"ACC","CCU"} 是序列集合中的第四个序列,那么指向 "U" 的键索引将为 {4,2,3}.
- 所有 DNA 和 RNA 序列字母都可以用 "MultiHydrogen" 键类型连接.
- 在肽序列中,并非所有键类型都适用于所有序列化学品. 以下键类型只能连接显示的肽字母:
-
"DisulfideBridges" C ↔ C, U ↔ U, C ↔ U "LactamBridges" D ↔ K, E ↔ K - 例如,BioSequence["Peptide","CGGGU",Bond[{1,5},type]] 中的 type 可以是 "DisulfideBridges" 但不能是 "LactamBridges".
- 基序序列的键也可以用点-括号表示法输入. 这种形式将序列的键表示为单个字符串,其中序列的每个字母对应于字符串中的那个位置. 键字符串的有效字符是句点 ("."),表示没有键或括号("(" 和 ")"),或尖括号("<" 和 ">")表示嵌套键对. 例如,字符串 "<((..>))." 适用于九个字母长的序列,等价于 {Bond[{1,6}],Bond[{2,8}],Bond[{3,7}]}.
- 通过 BioSequence[…]["prop"] 获得的 BioSequence 的属性 "prop" 包括:
-
"SequenceType" 作为 "BioSequenceType" 实体的序列类型 "SequenceString" 代表序列的字符串 "SequenceBondList" 序列中明确给定的键列表 "SequenceBondCount" 序列中明确给定的键数量 "SequenceLength" 序列的长度 "SequencePattern" 扩展退化性字母的字符串表达式 "AbbreviationSequence" 使用允许缩写的字符串表达式 "ChemicalList" 字面意义表示的化学实体列表 "ChemicalPatternList" 允许退化性字母表示的化学实体列表 "MolecularMass" 序列的分子质量 "MolarMass" 序列的摩尔质量 "HELM" 序列的 HELM 字符串 "Properties" 属性列表 - "ChemicalList" 或 "ChemicalPatternList" 都可以为序列的每个项给出特定化学物. 前者不支持退化性字母,而后者使用 Alternatives 来表示它们.
- 如果序列有退化性项,则其分子质量可能是 Interval.
- "HELM" 属性给出了 BioSequence 的大分子分层编辑语言 (HELM) 表示.
- BioSequence 可用的类型也可以通过用 "ExtendedBioSequenceType" 实体创建一个 EntityStore 然后进行注册(EntityRegister).
- 可定义以下 "ExtendedBioSequenceType" 属性:
-
"Alphabet" 在这个序列中允许的字母列表 "AlphabetRules" 从字母指定到特定化学物的一种关联关系 "BibliographicSource" 记录序列类型的外部标识符 "Caption" 在格式化的输出中序列上方的标题 "ComplementLetterRules" 定义互补操作的双向规则 "Icon" 显示在序列的格式化输出中的图标 "MolecularMassRules" 从字母指定到分子质量的一种关联关系 - 图标 "Icon" 可以图像或现存序列类型的规范名称的形式提供.
- "MolecularMassRules" 会通过 "AlphabetRules" 覆写化学物的分子质量并允许当没有化学物给定时计算质量.
- 只有当 bioseq 对应一个有效的 BioSequence 表达式时,BioSequenceQ[bioseq] 给出 True.
范例
打开所有单元关闭所有单元范围 (28)
属性和关系 (28)
BioSequence 提供了多个属性:
BioSequence 的类型是包含许多描述序列的属性的实体:
"MolecularMass" 和 "MolarMass" 属性适用于整个混合链:
对应 "BioSequenceType" 实体的 "Alphabet" 属性的给定类型的碱基字母:
BioSequence 的 motif 序列可以 Molecule 的输入形式提供:
混合链 BioSequence 也可以作为输入给 Molecule:
还可以向 Molecule 提供 BioSequence 集合:
使用 ConnectedMoleculeComponents 获得序列集合的单独分子:
SequenceAlignment 可在 BioSequence 的两个实例间进行对齐:
RandomInstance 可从退化性 BioSequence 中抽样完全指定的实例:
BioSequenceQ 可证实给定类型的 BioSequence 或有其他特征:
BioSequenceComplement 和 BioSequenceReverseComplement 求出 BioSequence 的遗传互补:
BioSequencePlot 显示 BioSequence 的示意图:
当将 "DNA"、"RNA"、"CircularDNA" 或 "CircularRNA" 类型的 BioSequence 转换为 Molecule 时,序列被解释为从 5' 3' 方向(正向):
当将 "Peptide" 或 "CircularPeptide" 类型的 BioSequence 转换为 Molecule 时,序列被解释为从 N 端到 C 端:
可能存在的问题 (4)
巧妙范例 (3)
文本
Wolfram Research (2020),BioSequence,Wolfram 语言函数,https://reference.wolfram.com/language/ref/BioSequence.html (更新于 2022 年).
CMS
Wolfram 语言. 2020. "BioSequence." Wolfram 语言与系统参考资料中心. Wolfram Research. 最新版本 2022. https://reference.wolfram.com/language/ref/BioSequence.html.
APA
Wolfram 语言. (2020). BioSequence. Wolfram 语言与系统参考资料中心. 追溯自 https://reference.wolfram.com/language/ref/BioSequence.html 年